2021-05-10
智源导读:中科院计算所卜东波团队近日于Nature Communications发表论文“CopulaNet: Learning residue co-evolution directly from multiple sequence alignment for protein structure prediction”,介绍一个新的神经网络架构CopulaNet,可从目标蛋白质的多序列联配直接估计出残基间距离,克服了传统统计方法的“信息丢失”缺陷;并以CopulaNet为核心开发了蛋白质结构“从头预测”算法和软件ProFOLD。在CASP13测试集上,ProFOLD达到了0.7的预测精度(以天然态结构和预测结构之间的TM-score为衡量标准),优于AlphaFold(约为0.5)。
CopulaNet和ProFOLD于2020年2月开发完毕,文章于2020年10月上传BioRxiv,并投稿至Nature Communications。在文章审稿期间,DeepMind公司于2021年公布了AlphaFold2的结果。ProFOLD目前虽然优于AlphaFold,但是与AlphaFold2相比尚有差距。卜东波老师团队正在努力改进ProFOLD,争取达到并超过AlphaFold2的水平。
关于蛋白质结构预测及其在生物学、药物研发方面的可能应用,智源社区与团队进行了深入访谈,探讨了这项工作的最新进展和未来挑战。

论文思路:所谓蛋白质三级结构,可以简单地理解成构成蛋白质的所有原子的空间坐标。蛋白质的三级结构可以从其残基间的距离精确地重建;就好比知道教室里同学们两两之间的欧式距离,就能确定出每位同学的平面坐标(在考虑旋转、平移、镜像等变换下是唯一的),残基共进化已经成为估计残基间距离的主要原则。大多数现有的残基共进化分析方法采用间接策略,即从目标蛋白质的多重序列比对(MSA)中提取一些手工的特征,比如协方差矩阵,然后利用这些手工提取特征推断残基共进化。
这种间接方法并不能充分利用 MSA 所携带的信息,从而导致相当大的信息丢失和残差距离估计不准。在这里,我们发布了一个端到端的深度学习框架(称为 CopulaNet) ,直接从MSA学习残基共进化。
研究结果表明,CopulaNet 能够有效地预测蛋白质三级结构。对于31个自由建模 CASP 13域中的24个域,我们的方法比现有先进方法获得了更高的预测精度。这项研究代表了端到端预测残基间距和蛋白质三级结构的重要一步。我们期望这里提出的方法可以得到进一步发展和应用,为理解蛋白质功能提供结构信息。
论文链接:Nature Communications,https://www.nature.com/articles/s41467-021-22869-8
预测服务器链接:http://protein.ict.ac.cn/FALCON/
预测软件源代码下载链接:http://protein.ict.ac.cn/ProFOLD/
访谈对象:
鞠富松,论文一作,中科院计算所博士研究生
卜东波,通讯作者,中科院计算所 研究员

左:中科院计算所博士研究生 鞠富松 | 中:中科院计算所研究员卜东波 | 右:中科院计算所博士研究生 孔鲁鹏
01 CopulaNet——从多序列联配直接预测残基间的共进化信息
CopulaNet的名字是怎么来的?请介绍一下CopulaNet的体系架构,具体是怎么想到用这个体系架构来进行蛋白质预测的?
卜:CopulaNet这个名字是北大统计系邓明华老师起的:用Copula表示“联合、联结”,指代“条件联合概率”。
这个工作的完成,主要是有几位学生愿意跟着我选择这个难度大、很冷门(虽然今年突然很热)的题目:第一作者是在读鞠富松同学,其他作者还有孔鲁鹏同学、已毕业的朱建伟同学(现在微软研究院工作)。
富松和鲁鹏都是纯粹觉得蛋白质结构好玩儿,纯粹从兴趣出发,不跟别人比发paper,心态比较好。
按照论文的逻辑,第一个就是说传统的方式是纯统计模型,是2011年 Chris Sander(哈佛大学计算生物学家)他们的做法,我觉得最重要的突破是从那开始的,从“共进化”开始。不过他们是从统计开始来的,统计就有一个很强的假设,假设在整个蛋白质数据中,找到同源的蛋白,同源指的就是在进化上比较相近的蛋白,把它都找过来;然后观察两个残基,比如第1个位置和第10个位置上的两个残基,它们之间的“共同突变”情况,根据共变情况推断残基间是否有接触,甚至进一步估计残基间的距离。
应用共进化分析技术能够预测残基间接触/距离,其背后的理论依据是:空间邻近的两个残基倾向于共同进化;因此反过来想,原则上可以利用残基共进化来估计残基间接触或距离。
常用的共进化分析技术主要有两种:假设目标蛋白质序列服从一个高维的正态分布,进而利用精度矩阵(协方差矩阵的逆)来表征残基间共进化程度,或者假设目标蛋白质序列由一个马尔科夫随机场模型(MRF)产生,进而用两体项表征残基间共进化程度。例如,AlphaFold和RaptorX都依赖于CCMpred预测的残基间接触,而CCMpred就使用了马尔科夫随机场模型。
现有共进化分析技术仍然存在一些不足,其根源在于:假设蛋白质序列服从一个高维的高斯分布,但这个假设是不成立的,我们也验证过。具体到精度矩阵来说,一个明显的缺点是“信息丢失”。
所以我们也在论文当中举了一个例子(图 1),显示了两个蛋白质,在序列和结构上都有显著差异,但是一旦按统计的模型算协方差矩阵,竟然完全相同;这就表明传统的统计模型有缺陷。接下来我们就做了一个新的模型,从而突破了传统统计模型的缺陷。

图1 基于高阶正态分布以及协方差阵方法的不足实例
这个实验方法主要的创新点在哪儿?
鞠:其实最主要创新点就是我们对于其他以前的方法,比如说AlphaFold 1,以及别人的模型,它的输入依然是用传统统计模型计算出的结果,然后输出是一个蛋白的残基距离。现在我们输入不用统计模型了,我们输入用原始的同源序列,这是我们跟别人相比最本质的一个差别。我们用的数据并不依赖于那些统计模型的结果。
我们开发的“从头预测“算法ProFOLD,将CopulaNet预测得到的残基距离转化为势能函数,并通过最小化势能函数得到蛋白质的三级结构。
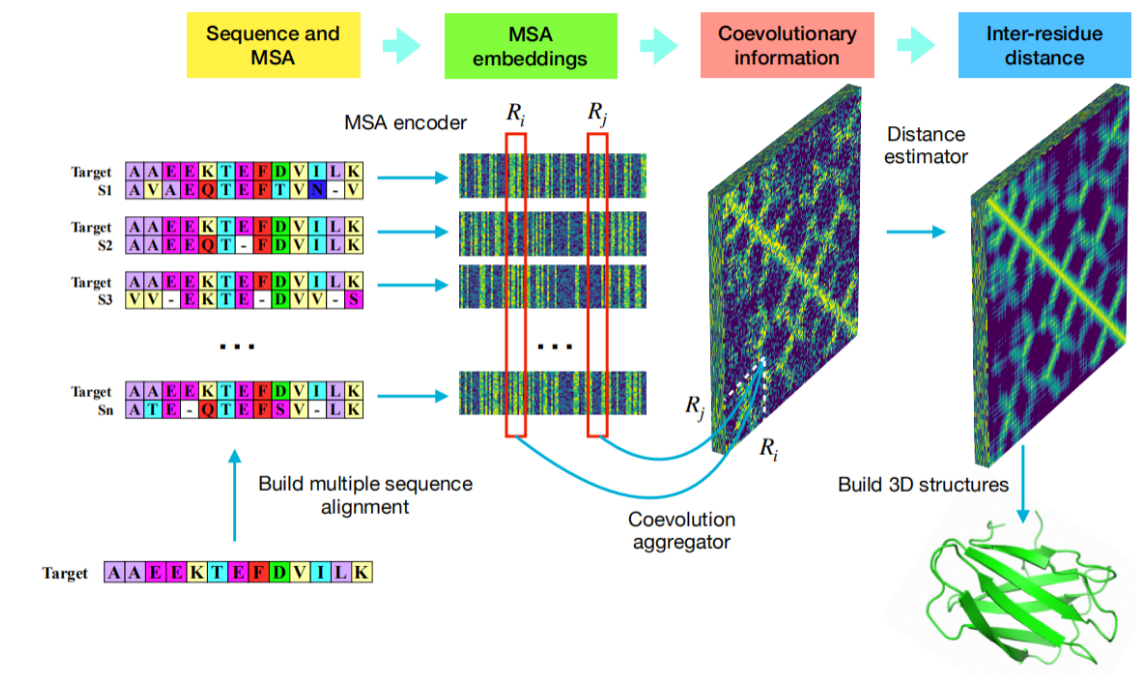
图2 ProFOLD预测蛋白质结构
它会更高效吗?还是会怎么样?
鞠:有几方面,一个是我们从更整体的机器学习的角度,越来越多人都是越来越接近于使用原始数据,而不是人工提取的feature。这是在其他领域也是很常见的事,总体思路就是,我们想引入更原始的数据,就尽可能减少损失信息。大家也都是这样,也可以说是end-to-end一个优势,更充分地利用我对数据的理解、数据所蕴含的根本性的信息,而不需要我们人为地做一些人工特征提取。
卜:传统方法都是基于统计模型的,他们基于统计模型产生的结果接着往下做。这样的话就相当于统计模型人工提的那些feature,出现问题的话,你后面就再也搬不回来了。所以我们现在绕过统计模型,end-to-end,直接从同源序列出发,估计残基之间距离。
如果绕过统计模型还算机器学习吗?
卜:算机器学习,因为机器学习不仅只有统计这一条路。
鞠:刚才说的统计模型其实就可以理解为人工提的特征,比如说NLP里面很早以前用的词袋模型(bag of words),或者CV里面很早以前用的金字塔,但现在都已经被抛弃了。我们刚才说的统计模型实际上指的是这种,人工提取一些先验的特征。
咱们的模型和AlphaFold相比有哪些优势劣势?
卜:我想描述成三个环节,就是AlphaFold目前已经有两代,AlphaFold和AlphaFold2,AlphaFold是基于统计模型出发的,相当于从 MSA 同源序列出发,先做统计模型,第二步估计距离,第三步是有了距离之后,模拟出三维结构,实际上是三步走。
我们做的ProFOLD是把前两步结合到一起,变成end-to-end的东西。AlphaFold2 相当于把这三步都给合到一块了,按照他的说法,细节还没有公开,大家都不知道。只是现在根据已有的信息,大家猜测是如此。从性能来看,AlphaFold是0.5,我们是0.7,AlphaFold2 大概0.92,指预测精度方面。
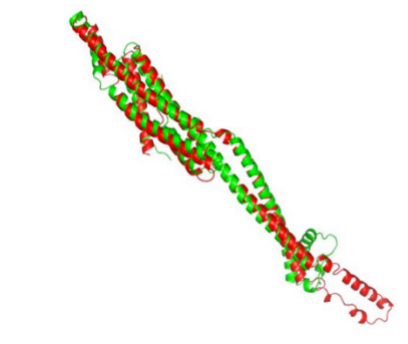
图3 用ProFOLD预测蛋白质三级结构示例。蛋白质:CASP13 FM类结构域T0950。红色:ProFOLD预测结构;绿色:天然态结构。TM-score=0.73
准确率是怎么来算的?比如 0.7、0.8。
鞠:类似一种结构上的相似度,越高越好。
卜:把两个三维的东西,经过一些旋转之后,最吻合的程度是怎么样?通常用TM-score、RMSD,或者GDT来度量。
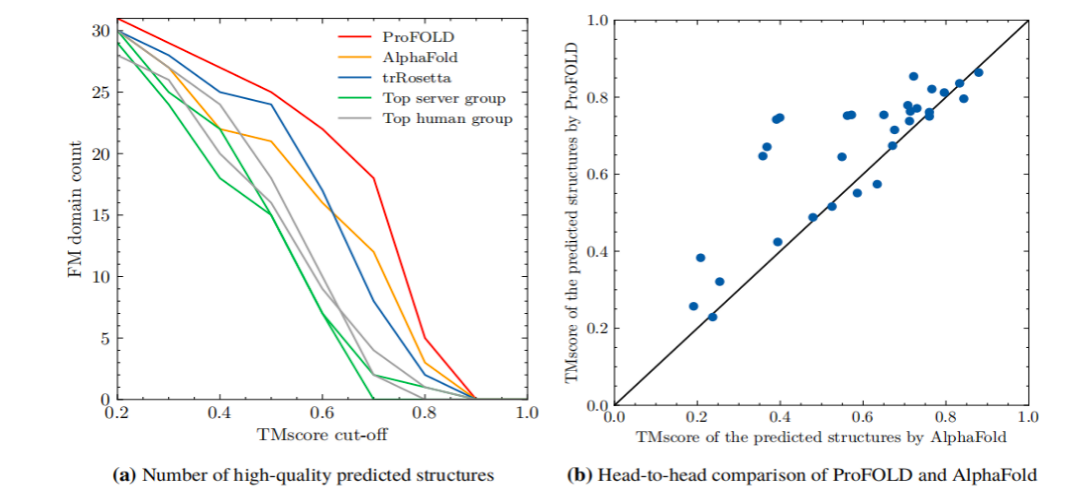
图4 用ProFOLD预测蛋白质三级结构。数据集:CASP13 FM类结构域
看到论文中说,残基少于500的目标蛋白在一台普通笔记本(Intel CPU 2.8 g Hz,16G 内存)上跑三个小时就可以完成结构预测过程,这个三小时是什么概念?
卜:500主要是说我们在笔记本上能跑多大,我们测过500残基的蛋白质,大概三个小时内即可完成预测。
训练和预测的 cost如何?
鞠:一般我们一个正常的四卡机工作站,训练10个小时左右,相比之下,像AlphaFold2 他自己说他训练了一个多月,我们的速度,还是可以接受的。
当然算力资源方面,可能他们100张卡、200张卡都是很轻松的,就不是可比较的一个计算力。
实际上他们方法是有创新的,Jump曾经在CASP14 meeting里做过一次报告,透露了只言片语,但具体的创新点我们并不知道。David Baker也说报告讲得不详细。
ProFOLD现在预测结果有没有验证,有没有实际的应用?
卜:验证就是CASP-13测试集,大家都公认的一个国际比赛的测试集,比较客观、公正。
这样的话我们在上面性能是超过AlphaFold,trRosetta等,比他们都要好。实际应用方面,我们在2015年开发了蛋白质结构预测服务器FALCON,业界同行在用,截止到目前,已经预测了105,101个蛋白质的结构。现在包括大连化物所等都在用。
现在另一个博士生孔鲁鹏和富松合作,把FALCON升级成FALCON2,集成了“从头预测”算法ProFOLD,以及“有模板预测算法”ProALIGN。ProALIGN是鲁鹏(也是本文的作者之一)为主开发的,文章发表于生物信息学会议RECOMB上。
02 寻找同源的可计算定义
这项工作的主要困难点在哪里?
鞠:有几方面,数据多,计算量大,再就是定义不是很清楚。
蛋白质数据库都是公开的,现在应该有十几万个已知结构的蛋白,各方面来源的序列,如果总共算起来的话,大概有几个billion这么大。蛋白的序列其实是很多的,但我们已知结构的数据就大概有十几万,这两个量级还是差挺多的。
所以寻找同源序列的困难点主要是耗时?
鞠:寻找同源这个问题主要不是耗时,而是我们无法确定它是不是同源,这个问题,现在还没有说有一个比较现代的机器学习方式来做,都是用非常老的统计模型来做这件事。
这件事本身问题的定义也比较模糊,或者说我们的需求也比较模糊,同源序列的定义现在应该是整个蛋白里定义最不清楚的一块,它本身是没有什么特别严格定义的。
定义模糊指的是?
鞠:就像我们描述说,同源是指两个蛋白是由同一个祖先进化来的,但我们怎么知道他们是不是同一个进化来的,进化过程我们是不清楚的。
卜:怎么样是同源,这件事情非常好描述;但是实际上计算的时候,无法定量计算。就跟语言学一样,我们各地的方言,可能一开始是从一个最古老的我们原始的文字来的,但是后来为什么现在那么多语言。
打个比方,我们汉语跟英语的差异非常大,但是汉语与日本话比较接近。日本人可能借鉴了我们很多片假名里的东西,这个从进化来说我们是很近的,但你要具体的来刻画汉语跟日本语之间差异,这就麻烦了,不好有个严谨的可计算的定义。所以在计算语言学里,通常不考虑语言之间的进化关系。
总结来说,整个问题的瓶颈应该就是在这一块——同源的可计算的定义是什么。
03领域应用:药物小分子与蛋白质的作用位点
选择预测蛋白质结构这一领域的初衷?
卜:我是从2006年开始,到加拿大李明教授实验室访问,和李明老师、李帅成(现在香港城市大学教授)、许锦波(现在芝加哥丰田研究所,RaptorX的开发者)一起开展蛋白质结构预测研究。
我做蛋白质结构预测,有两个动机:一是兴趣,在这个驱动力上我跟富松很像,都是首先觉得好玩儿。我觉得很多蛋白质结构长得很好看。第二,我觉得关键是,这个问题在我们选题来说它是有标准答案的,就是蛋白质折叠的构象,天然态构象是基本上确定的,基本上是由序列确定的,是有标准答案的。不像是其他的一些学科,它可能没有标准答案这是我选择领域中最重要的一个出发点。我有个可学习的目标,我原来做文本分类的时候,你看文本分类,这篇文章是属于政治类还是经济类?标准答案也不标准,是请了一些专家人为划分的;往往公说公有理,婆说婆有理。
实际应用落地方面,未来的前景怎么样?
卜:比如说制药,药物那一块很重要的一件事情就是,药物是个小分子,我们先说化学药不说生物药。化学药它是个小分子,我们就想知道小分子作用在哪个蛋白上,要想知道这个事情,你首先得知道蛋白质的结构,这是他们非常关注的核心问题。进一步,第二个问题就是作用在蛋白质上,具体的位点是哪一块,这是他们关注的两个基础性的问题。
第三步,衡量这个小分子的药效,小分子跟我这个蛋白的结合能力是怎么样?这是进一步的一个问题,在这些基础上知道蛋白质结构是很重要的、技术性的环节。
比如中科院大连化物所梁鑫淼老师建立的中药科学中心,用液相色谱和质谱技术把中药材的成分都鉴定出来,鉴定出到底有哪些小分子;在这方面梁老师团队,包括郭志谋、闫竞宇、周晗、叶贤龙等老师,做了非常好的工作。进一步的工作是确定小分子作用机理,比如和蛋白质之间的作用位点;我们正在和梁老师团队开展密切的合作。
04 CS人要向物理学家学习「求知」精神,而不仅仅是刷榜
全国有哪些团队也在做类似的事情吗?
卜:国内在蛋白质结构预测相关领域有多支团队开展研究工作,大致可以分作3个层面
(1)从生物物理、化学角度对蛋白质折叠机理和蛋白质设计等方面的探索:如中国科技大学施蕴渝/刘海燕团队、北京大学来鲁华/宋晨/裴剑锋团队、朱怀球团队、北京工业大学王存新团队、中科院理论物理所郑伟谋团队、中国医科院基础医学研究所蒋太交团队等在蛋白质折叠机理、分子动力学模拟、蛋白质结构预测方法和蛋白质设计方面有长期的积累;吉林大学田圃团队提出了“广义溶剂自由能”理论,设计了“神经网络力场”;华中农业大学邓海游团队改进能量函数进行全原子结构优化;清华大学龚海鹏团队在蛋白质能量函数设计、局部结构预测、Ab Initio算法设计等方面有突出成果.
(2)从计算机算法和机器学习角度对蛋白质结构预测算法的探索:如清华大学曾坚阳、崔雪峰团队使用深度学习技术进行蛋白质结构预测和电镜信号分析;中国人民大学龚新奇团队、同济大学黄德双团队、南京大学周志华团队、浙江工业大学张贵军团队应用机器学习算法于蛋白质超二级结构预测、空间结构预测等方面有良好的积累;上海交大沈红斌团队应用机器学习技术预测蛋白质残基相互作用;南开大学杨建益团队同时预测残基间距离和相对角度,开发了软件trRosetta;香港城市大学李帅成团队和我们有密切合作,在蛋白质结构预测的Ab Initio算法ProFOLD、threading算法ProALIGN、局部结构预测、能量函数设计等方面有较好的成果。
(3)应用蛋白质结构解决药物设计、分子对接、DNA绑定位点等实际问题:华中农业大学刘融团队预测蛋白质与DNA的结合位点;华东师范大学戚逸飞团队基于结构预测膜蛋白突变之后的稳定性;上海海洋大学徐舒坦团队研究蛋白质的柔性对接;华中科技大学黄胜友/肖奕团队采用FFT技术分析蛋白质-DNA结合位点处的空间构型;中山大学杨跃东团队利用深度学习技术对蛋白质和小分子进行结构编码,进而研究药物筛选和智能设计。
国内研究团队也多次参加CASP比赛,包括中科院生物物理研究所蒋太交团队的Jiang-Server、上海交通大学沈红斌团队的Shen-group、腾讯公司的tFold,以及我们所在的FALCON团队。
我们团队是从2006年开始做的;从2008年就开始和理论物理所的郑伟谋老师一起合作,郑老师也是这篇文章的共同作者之一。
物理所的贡献主要是体现在哪些方面?
卜:理论物理所的郑伟谋老师是一直跟我们密切合作,从2008年就开始了 。郑老师从物理方面提供很多insights,对我们有非常多指导性的意见,最重要的是一些“生物学洞察”。因为蛋白结构它本身是个生物物理的问题,就是蛋白质为什么会折叠成这样的?他是从这个机理的角度来考虑的。
和物理所的老师是怎么跨学科协作的,不同学科之间交叉会有什么样的感受?
卜:我们是经常听不懂就多问。不同学科的差异很明显。计算机这边 engineering的味道太重了,就是我们光讲性能,不问为什么。物理学是要知道为什么,想知道新的knowledge,这是非常大的差异。我们在郑老师的鞭策下,我们努力地改正了很多的计算机这边的思维定式。
光讲刷分刷榜是不好的。UCLA的朱松纯老师也写过一篇长文,明确反对计算机界,尤其是CVPR等一些会议盲目刷榜的问题。
我最近也在写篇文章,对比一下物理学家、统计学家以及CS人,看待同一个问题的不同视角,以及解决同一个问题的不同入手方式。
有哪些具体的insights?
卜:比如说我们这个工作里面最重要的就是一个共进化假设。两个残基,如果是他俩离得比较近,它俩就相互作用的话,那么它俩在进化上你变我也变。这是2011年 Chris Sander文章中最核心的一点。这也是整个领域比较基础性的假设。
我们在同源序列上就能观察出来,反过来只要观察到这两个经常一块变,它俩之间应该距离比较近。我们整个方法,甚至最近整个领域的重要进展,都是从这里来的。
在进化树上,不管在哪个分支上,这两个残基都一起变化的。他俩在空间上很有可能就绑定在一块了,就是这么一个假设。那反映到结构上,进化到底该如何认识,郑老师给我们很多指导。
对于蛋白质折叠过程,郑老师把他的认识总结成4句话,是“精英绑架、层次折叠、强弱搭配、弱是必须”。

图5 郑伟谋老师总结的对蛋白质折叠过程的认识
郑老师指导我们进行探索,进而郑老师总结了对蛋白质折叠机理的一系列认识。这是我们非常佩服他的地方,我们很多工作当中潜移默化都在受他的影响。比如局部结构和整体结构之间的关系;能量项那么多,哪种先起作用、哪种后起作用等等。这些观点和认识,对于我们设计预测方法,是非常重要的。
您认为跨学科协作的启示是什么?
卜:我的感觉一定是要彻底地解放自己,不要把自己太局限于自己熟悉的领域,一定要以极大的兴趣去了解另外一个领域,这样才能做好交叉学科。
绝不能说,我们是计算机出身的,是学算法的,就认为自己就是个搞计算的,要对另外一个学科有好奇心和兴趣。现在我们也在设计生物学的实验,不要把自己太局限到自己是个计算机系的人,这是第一点。
第二点我觉得不同学科有不同的特色,像物理学家就有非常值得我们借鉴的特色。
物理学家有非常强烈的想要知道背后原因的动机,比如费曼,再比如写了《生物是什么》的薛定谔,影响了很多人,包括发现DNA双螺旋的Watson和Crick。而反观计算机系的人,尤其是近期搞AI的人,常常是不去追求why的。我觉得我们搞计算机的,要向物理学家学习这种“求知”的精神,而不是仅仅刷榜。
附:蛋白质结构预测服务器FALCON2 http://protein.ict.ac.cn/FALCON2
论文作者孔鲁鹏和鞠富松开发了蛋白质结构预测服务器FALCON2,集成了“从头预测”算法ProFOLD和“有模板预测方法”ProALIGN(第一作者孔鲁鹏,文章发表于RECOMB2021),为学术界提供蛋白质结构预测服务。
图6 FALCON2蛋白质结构预测服务器
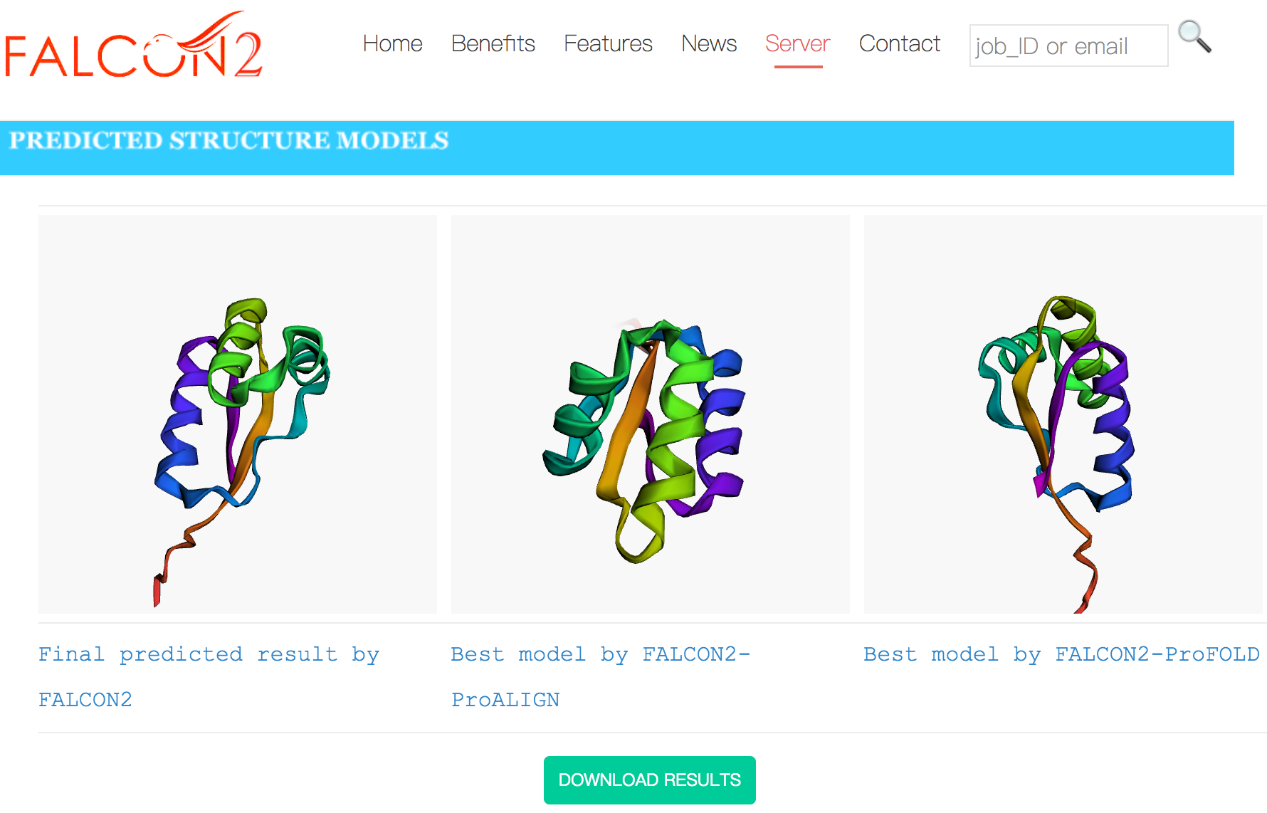
图7 FALCON2蛋白质结构预测服务器预测实例。蛋白质:1ctfA
(转自智源社区)